高阶菌群标志物怎么筛选?试试「多组学联合标志物+验证实验」!
【2020-08-03】
在六月份的公开课中,我们谈过了目前肠道菌群研究可以大致分为五个方向其中疾病标志物筛找是一个非常重要的研究方向,也具有十分重要的临床意义。本文将从两篇“结直肠癌疾病标志物筛找”的案例中,探讨胃肠道疾病领域的疾病标志物筛找研究如何进行实验设计和结果分析。
首先,可从下图中了解肠道菌群疾病标志物的筛找通用流程。
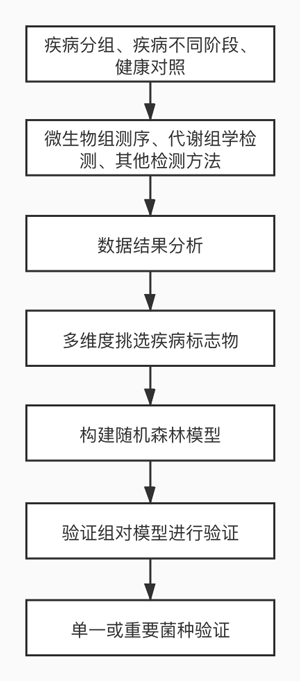
疾病标志物筛找通用实验设计流程
案例1
大规模独立宏基因组+代谢组实验筛找疾病不同时期标志物

研究内容:宏基因组和代谢组分析揭示肠道菌群明显的结直肠癌阶段性特异表型
发表时间:2019年6月
发表期刊:Nature medicine(IF:36.13)
结直肠癌可分为遗传性结直肠癌和散发性结直肠癌,在散发性结直肠癌的发生过程中,会先形成息肉样腺瘤,然后发展成为粘膜内癌,最终成为恶性肿瘤。本研究对千余名健康人和不同阶段的结直肠癌患者进行研究,利用宏基因组和代谢组检测的方法,以探究粪便微生物和代谢产物在不同结直肠癌发展阶段性特异表型,筛找出疾病标志物,为CRC发展阶段的诊断治疗奠定理论基础。
实验设计
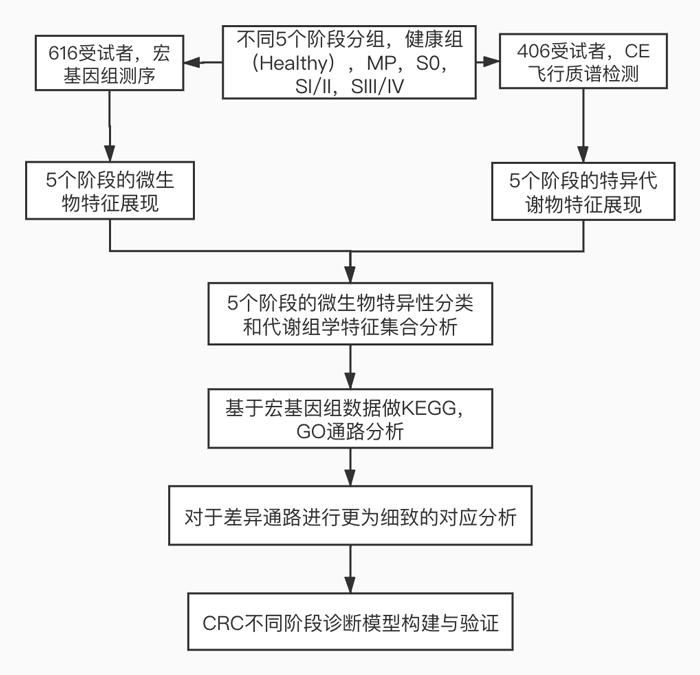
实验设计流程图
CRC不同阶段诊断标志物筛找与建模
本研究利用在不同阶段CRC病人中得到的结果,分别用差异菌种、KO基因、代谢产物和三者的组合构建随机森林和LASSO逻辑回归分类,以达到利用标志物区分健康对照与不同CRC阶段(S0、SIII / IV)的目的。模型结果显示,在S0、SIII/IV分类中,三者组合参数模型的分类分辨率优于单个因素,区分 CRC 患者 S0和SIII/IV期的AUC值分别为0.78和0.85,具有较好的区分效果。
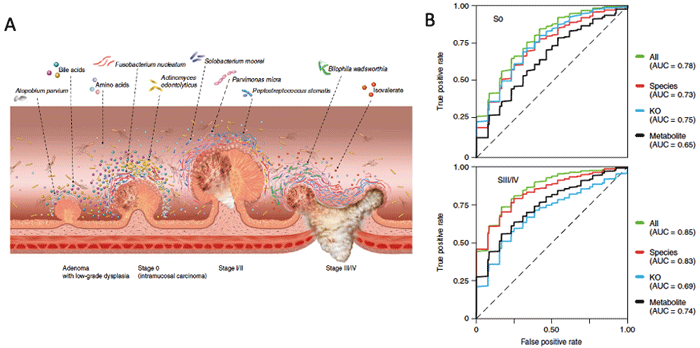
A.在不同CRC疾病阶段特异的微生物与代谢物
B.随机森林ROC曲线
文章评述
本研究的目的在于筛找结直肠癌不同发展阶段的疾病诊断标志物。通过利用宏基因组测序和代谢组学检测的方法,鉴定出了在CRC不同阶段微生物与代谢组的变化情况。最后作者采用菌种基因(specis)、功能基因(KO)和代谢产物(metabolites)三个指标构建随机森林的方法来判断疾病不同阶段,这对CRC的早筛有着极为重要的意义。
本研究是胃肠道疾病研究领域筛找疾病标志物方向上的一篇很具有参考意义的文章,能够给我们的启发有以下几点:
01 实验规模大
在胃肠道领域筛找疾病标志物的研究中,由于研究较为成熟,对样本规模有较高要求。本研究对600多个样本进行宏基因组测序,400多个样本进行代谢组学检测,大量样本的实验在筛选疾病标志物上更加具有说服力。
02 微生物+代谢组联合分析
双组学联合分析的方法能够多一个维度探究问题,并深入到疾病的因果关系层面。
03 组合参数,提高区分能力
本研究在构建随机森林模型时,尝试把菌种、KO基因和代谢产物三个单独的参数组合起来,提升了分辨率,值得参考。
04 强大生信分析能力
非常细致的生信分析,信息量庞大的生信分析图,凸显了课题组生信分析实力。
案例2
利用宏基因组测序+qPCR验证方法筛找疾病诊断标志物

研究内容:通过比较健康家庭患者的粪便菌群,建立结直肠癌的高精度生物标志物
发表时间:2020年1月
发表期刊:Gut microbes(IF:7.74)
本研究目的是筛找结直肠癌的早筛诊断标记物,采用宏基因组测序方法对52个结直肠癌病人和55个健康对照进行研究,并挑选出22个微生物作为标志物构建模型,后再用46个CRC病人和40个健康人对模型进行验证,最终筛选出了3株具有良好疾病诊断潜力的菌种,为结直肠癌的早筛奠定了理论基础。
实验设计
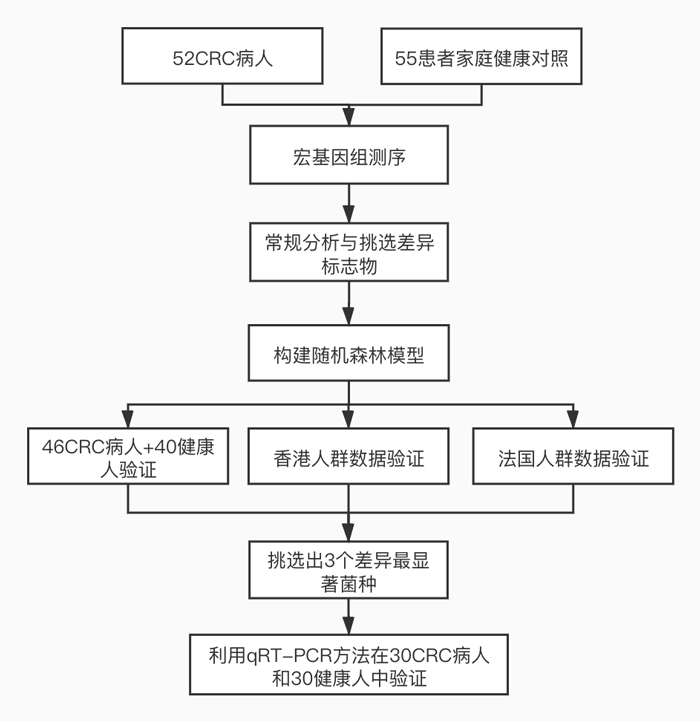
实验设计流程图
CRC确诊标志物筛找、建模与验证
通过对宏基因组的数据分析,挑选了22个和CRC显著相关的微生物功能基因,其中一半基因来源于厚壁菌门。这些功能基因涉及到群体感应、短链脂肪酸代谢、基因信息处理、DNA服务与修复等。利用此22个基因构建随机森林模型,分别对验证组(46CRC,40健康人)、香港地区数据(74CRC,54健康人)和法国地区数据(53CRC,88健康人)进行验证。其中验证组、香港组、法国组的AUC值分别为0.905、0.811、0.886。
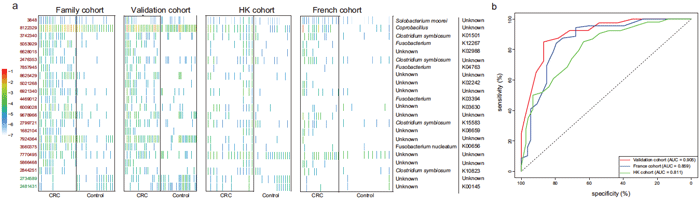
A.22个微生物功能基因在不同组别中丰度信息
B.随机森林对验证组、香港数据和法国数据ROC图
从22个基因中选取3个最具有诊断潜力基因,并设计qPCR探针,在30个CRC和30个健康人队列当中进行检测,其中2个来源于Coprobacillus和C.symbiosum的基因在CRC和健康人群中显著差异,构建随机森林模型,AUC值分别为0.930和0.935。
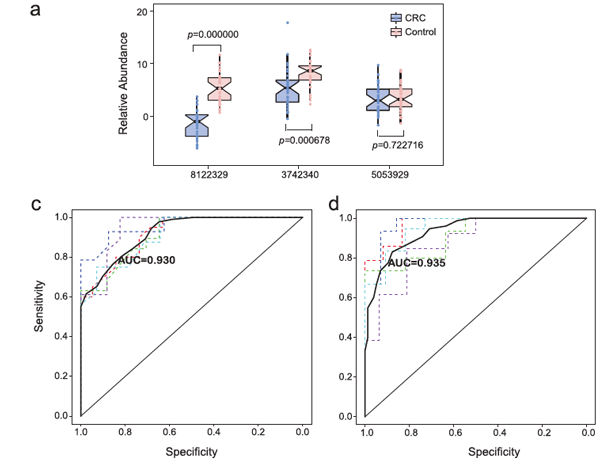
A.3基因在分组间相对丰度
C&D.2个显著差异基因构建随机森林模型
文章评述
本研究目的是筛找结直肠癌早筛标志物。在实验过程中,其运用的方法和思路非常经典,值得大家学习和参考。
01 试验设计:在筛找标志物的发现组中,研究选择CRC患者,而健康对照也选用他们的家人。这一点设计非常巧妙,既简化了对照志愿者的征集工作,又可在一定程度上保持对照和实验组在饮食条件上保持一致。
02 构建模型后进行验证:本研究在构建模型后,不止用验证组做验证,还采用了其他地区人群的数据,起到了扩展验证面的作用,值得借鉴。
03 单一菌种的验证:根据验证结果挑选出了区分效果最优的三个菌种,用qRT-PCR的方法在另一个验证组中做验证。
总结
通过总结上述两篇文章及多篇胃肠道领域筛找疾病标志物研究,提炼出了胃肠道领域筛找疾病标志物的研究趋势:
(1)实验规模再度提升,纯数据分析更是如此:纵观近期发表的疾病标志物筛找的实验,实验规模和样本量都是比较大的。一般自主实验的研究样本量至少也要达到几百之上,而纯数据分析的实验,通过集合已发表文章的数据进行分析,因此样本数量至少上千。
(2)标志物的寻找从菌属到菌种再精确到菌株,方法从16S向宏基因组转变:从上两篇文章和近期发表的高分文献当中即可看出,微生物测序的方法已经从16S测序转变为宏基因组,因为宏基因组测序可以将菌种分类做的更好,同时得到代谢功能基因的信息,其也可作为诊断标志物。
(3)菌种+功能基因+代谢产物,构建模型时使用多组学标志物分辨率更高:众多疾病标志物筛找的实验中都尝试将多组学的标志物结合在一起建模,通常来说,这样的方法不仅能丰富文章内容,也可将模型分辨率提升。
(4)构建模型后,需要设置验证组对模型进行验证:对于肠道菌群标志物筛找的研究来说,通常需要设置验证组,来验证由实验组建立出来的模型。如果有条件,也可分离单菌种,用小鼠模型去验证因果关系或进行更深一步的实验。
参加流程
● STEP1:扫码填写申请表。
● STEP2:工作人员3个工作日内电话沟通,了解项目详情。
● STEP3:阅微基因与中科院微生物所专家委员会评审,5个工作日内通知,通过则签署合作协议。
● STEP4:客户需在规定时间内完成送样,开展项目合作。

扫码填写项目征集表,轻松完成申请,
5个工作日反馈申请结果







